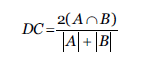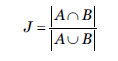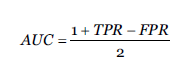1. Introducción
Las malformaciones en la estructura cerebral se refieren a alteraciones en la forma, tamaño o posición de la estructura cerebral, lo cual puede afectar la función neurológica de una persona y su salud en general (Aronica et al., 2012). Estas alteraciones pueden ser desarrolladas en una etapa embrionaria (malformaciones congénitas) o adquiridas a lo largo de la vida. Los cambios estructurales congénitos se deben usualmente a una disrupción en el desarrollo normal de la etapa embrionaria o fetal (Sarma & Pruthi, 2023), mientras que las malformaciones adquiridas corresponden usualmente a enfermedades, lesiones, circunstancias ambientales u otras causas externas (Peruzzo et al., 2016). En general, este tipo de condiciones conlleva a enfermedades como epilepsia refractaria causada por displasias corticales, consideradas como la causa principal de esta enfermedad (Sánchez Fernández et al., 2021), Alzheimer debido a contracciones en regiones del cerebro (Ito et al., 2019), esclerosis múltiple por pérdida de volumen cerebral (Clèrigues et al., 2023), microangiopatía causada por los espacios de Virchow-Robin (Rashid et al., 2022) y en el caso de malformaciones como las de Chianni, pueden aparecer múltiples síntomas como desequilibrio, tinnitus, dificultad para tragar, palpitaciones, apnea del sueño, debilidad muscular, fatiga crónica y tinciones dolorosas (Kumar et al., 2011).
El estudio de la estructura cerebral ha sido impulsado por el análisis de imágenes médicas. Específicamente, se han utilizados neuroimágenes para realizar estudios cuantitativos acerca de la estructura y funcionalidad del sistema nervioso central. Las imágenes por resonancia magnética (MRI), son uno de los tipos de neuroimágenes ampliamente utilizadas, estableciéndose como una herramienta valiosa para el diagnóstico de enfermedades neurológicas, y una gran ayuda al momento de evaluar la morfología cerebral (Castillo-Carranza et al., 2022; Hyman, 2003), permitiendo tanto la evaluación de la patología asociada a síntomas clínicos o cognitivos, y un diagnóstico diferencial (Zubrikhina et al., 2023). Mediante MRI es posible diferencias estructuras del tejido cerebral como la materia blanca, materia gris y líquido fluido-espinal en tareas como clasificación, identificación y segmentación (Clèrigues et al., 2023; Jiang et al., 2023). La línea base para estas tareas se da de forma manual bajo el criterio y experticia de médicos especializados como radiólogos al ejecutar una inspección visual de las imágenes. Sin embargo, realizar este proceso de forma manual supone retos en cuanto al manejo de grandes bases de datos, sesgos y equivocaciones asociadas a la diferencia de contrastes entre imágenes que suponen un reto incluso para los expertos dada la falta de criterios consistentes (Ito et al., 2019). En los últimos años, la segmentación de MRI cerebral ha sido conducido por técnicas de aprendizaje profundo. Este método consiste en la implementación de redes neuronales artificiales con gran volumen de capas y neuronas, las cuales logran extraer una jerarquía de características de imágenes de entrada sin formato. Algunos de los algoritmos de aprendizaje profundo conocidos son codificadores automáticos apilados, máquinas de Boltzmann profundas, redes neuronales profundas y redes neuronales convolucionales, siendo las últimas las más comúnmente aplicadas a la segmentación y clasificación de imágenes (Akkus et al., 2017; Toscano et al., 2023).
Se ha encontrado en el estado del arte un amplio rango de aplicaciones de algoritmos de aprendizaje profundo en la segmentación de estructuras en MRI. En (de Brebisson & Montana, 2015), propusieron una arquitectura de red neuronal profunda para segmentación automática, donde obtuvieron resultados promisorios de validación entrenando grandes redes, compuestas de decenas de millones de parámetros, con una cantidad relativamente pequeña de datos. En (Somasundaram & Kalaividya, 2016), emplearon un algoritmo de deconvolución Richardson-Lucy (RL) para mejorar la detección de límites y la calidad de imagen, tomando funciones de difusión de puntos tipo Gaussiano y extraer porciones del cerebro de imágenes de resonancia magnética coronal T1-W; también utilizaron los procesos de intensidad de umbralización, componente conectado y operaciones morfológicas para extracción cerebral. Este proceso ayudó a identificar el límite cerebral mejor que otros métodos. Para (Chang & Hsieh, 2017), surgía un reto más grande; separar el tejido cerebral de tejido no cerebral, ya que representaba un problema mayor al realizar la segmentación del cerebro, por ello, presentaron un algoritmo de extracción de cráneo basado en la combinación de análisis de características de textura de imagen y morfología matemática. Este algoritmo obtenía dos mapas de características de textura, donde uno correspondía a una máscara cerebrales y el otro a una máscara no cerebral. Posteriormente se realiza una serie de operaciones morfológicas para extraer el cerebro. En (Avants et al., 2011), presentaron Atropos, un algoritmo de segmentación de código abierto multivariado de n clases basado en Insight Toolkit (ITK). Para el desarrollo de este método, emplearon algoritmos de maximización de la esperanza (EM) con el modelado de las intensidades de clases, basadas en mezclas finitas paramétricas o no paramétricas. También, incorporaron mapas espaciales de probabilidad previa, mapas de etiquetas anteriores o modelado de campo aleatorio de Markov (MRF). En (Billot et al., 2020), presentaron un método de aprendizaje profundo denominado “SynthSeg”, donde aprovechaban un conjunto de segmentaciones de entrenamiento, para generar escaneos sintéticos de contrastes muy variables sobre la marcha durante el entrenamiento. Estos escaneos se producían utilizando el modelo generativo del marco de segmentación bayesiano clásico, con parámetros muestreados aleatoriamente para apariencia, deformación, ruido y campo de sesgo, y como resultado, obtuvieron una segmentación exitosa de cada contraste en los datos, con un rendimiento ligeramente mejor que la segmentación bayesiana clásica y tres órdenes de magnitud más rápido. Finalmente, en (Chen & Merhof, 2018), presentaron “MixNet”, una red neuronal convolucional profunda 2D semántica para segmentar la estructura del cerebro en imágenes de resonancia magnética multimodal, reemplazando la capa convolucional tradicional por una capa convolucional dilatada, evitando el uso de capas agrupadas y capas deconvolucionales, reduciendo el número de parámetros de la red. Al final obtuvieron un coeficiente de Dice general de 84.7 % para materia gris, 87.3 % para sustancia blanca y 83.4 % para líquido cefalorraquídeo, con solo 4 sujetos como entrenamiento.
En el presente trabajo se presenta una red neuronal convolucional (CNN), basada en la arquitectura U-Net para la segmentación de imágenes y volúmenes de MRI de cerebro, obteniendo como resultados imágenes segmentadas con etiquetas correspondientes a: fondo, materia gris, materia blanca y fluido cerebroespinal. Posteriormente se comparan mediante cuatro métricas de desempeño los resultados obtenidos respecto a otros métodos de segmentación de MRI usados como Dipy y el método FAST de la librería FMRIB Software Library (FSL). Los resultados muestran que la segmentación de imágenes MRI empleando redes convolucionales, específicamente las redes U-Net 2D y U-Net 3D, son más efectivas que los demás métodos implementados, ofreciendo un mejor desempeño para segmentar las estructuras cerebrales sin necesidad de recurrir a módulos o software externos.
2. Metodología
La Figura 1 presenta el esquema metodológico para el análisis de MRI, el cual comprende los siguientes pasos. La realización de los estudios clínicos para la adquisición de las imágenes, la conformación de un banco de datos sobre condiciones específicas a tratar, es decir, las enfermedades o deformidades existentes en los pacientes, y los análisis realizados por el equipo médico experto que determina las condiciones clínicas que luego son utilizadas como etiquetas para entrenar los modelos. En este trabajo, se propone en la etapa del aprendizaje de máquina el uso de modelos de aprendizaje profundo, específicamente la red U-Net que se ha mencionado previamente. Con el entrenamiento de los modelos, luego se procede a validar con el conjunto de datos de prueba. El objetivo de este estudio es la segmentación precisa del área focal (materia gris y materia blanca), evitando resaltar otras zonas. Finalmente, en el esquema general se contempla la evaluación del modelo, en este caso se utilizaron métricas que determinan la similitud entre la segmentación del modelo y la segmentación dada por el experto.
2.1. Métodos de adquisición de imágenes médicas
Existen diversas tecnologías de adquisición de imagen médica, desde la obtención de imágenes empleando rayos X hasta llegar a los modernos dispositivos multimodales. Dentro de estas tecnologías se pueden encontrar seis de ellas como las más reconocidas a nivel general, empezando por las de ultrasonido, radiografías, tomografías, MRI, Gammacámara y finalmente la fluoroscopia.
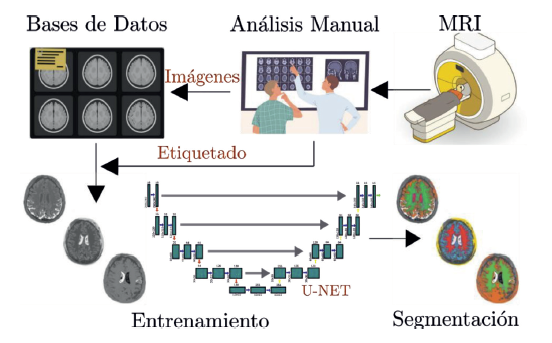
Figura 1 Estructura metodológica propuesta para la segmentación de imágenes de resonancia magnética basado en aprendizaje profundo.
La MRI se destaca al ser una herramienta muy potente para el diagnóstico anatómico debido a su alta resolución espacial y a que no es invasiva; lo cual la hace un examen por excelencia para el estudio de imágenes médicas. La MRI permite generar imágenes anatómicas tridimensionales en escala de grises, lo que es una gran ventaja a la hora de realizar procesamiento de estas (Hyman, 2003).
2.2. Red neuronal convolucional (CNN)
Las redes neuronales convolucionales (CNNs, por sus siglas en inglés) son un tipo de redes neuronales artificiales utilizadas frecuentemente para el análisis y reconocimiento de imágenes (Anwar et al., 2018; Ib et al., 2022; Liu Qing and Zhang, 2017). Estas redes son altamente efectivas en tareas de visión artificial, tales como clasificación y segmentación de imágenes, donde a diferencia de las redes neuronales convencionales que utilizan operaciones lineales entre las capas para obtener representaciones de las entradas, las CNN utilizan capas convolucionales, y a medida que la entrada pasa por cada una de las capas, estas van aplicando filtros a la imagen que logran abstraer información no lineal de los pixeles analizados, generando características discriminatorias.
Las redes neuronales convencionales son totalmente conectadas o fully connected (FC), es decir, todas las neuronas de una capa oculta están conectadas a todas las neuronas de las capas que le preceden y suceden (Alzubaidi et al., 2021). Este proceso genera una cantidad enorme de parámetros (también conocidos como los pesos de la red), debido a que cada capa que tenga la red va a tener conectada cada neurona entre sí, y a su vez, estas neuronas de cada capa se conectarán con cada píxel de la imagen, lo que supondrá un problema cuando se requiera entrenar una red con un volumen considerable de imágenes, ocasionando una inviabilidad por la enorme cantidad de pesos que se obtendrían.
Las redes neuronales convolucionales surgieron como una alternativa de solución a lo anterior. Estas redes cuentan con capas convolucionales y de agrupación que se alternan y finalizan con unas capas FC. Una capa convolucional consiste en filtros o kernels de tamaño f × f × c cada uno, donde c hace referencia a la profundidad o el número de canales (3 para imágenes a color, 1 para imágenes en grises). Adicionalmente se suele introducir una capa de agregación o pooling a la salida de una convolución que reduce el tamaño de las imágenes realizando la correspondiente operación de agrupación, comúnmente máximo o media (“MaxPooling” o “AvgPooling”) (Alzubaidi et al., 2021).
Dentro de las redes neuronales convolucionales, se han logrado desarrollar múltiples arquitecturas de redes CNN que han obtenido resultados destacados en diferentes áreas. Algunas de ellas son la LeNet (LeCun et al., 1998), AlexNet (Krizhevsky et al., 2012), VGGNet (Simonyan & Zisserman, 2014), U-Net (Ronneberger et al., 2015), GoogLeNet (Szegedy et al., 2015), entre otras. De estas, se empleará la arquitectura U-Net (Siddique et al., 2021) para desarrollar el algoritmo de segmentación de imágenes y volúmenes médicos.
2.3. Arquitectura U-Net
La red U-Net es una red convolucional para segmentación rápida y precisa de imágenes médicas. Existen dos arquitecturas U-Net; la primera arquitectura es la red U-Net 2D, compuesta de una ruta de contracción que sigue la arquitectura típica de una red convolucional; convoluciones 3 × 3, unidades lineales rectificadas (ReLU), operaciones MaxPooling 2D y una ruta de expansión que consiste en un muestreo ascendente del mapa de características, seguido de convoluciones 2 × 2 (“convolución ascendente”) que reduce a la mitad el número de canales y convoluciones 3 × 3, cada una seguida de una ReLU. En total, la red tiene 23 capas convolucionales (Ronneberger et al., 2015). La segunda arquitectura es la red U-Net 3D, en esta se usará el volumen completo (o subvolúmenes) y se usarán capas convolucionales, y operaciones de agrupación máxima tridimensionales (MaxPooling 3D), tanto para la ruta de contracción como para la ruta de expansión (Çiçek et al., 2016; Ronneberger et al., 2015).
2.4. Otros métodos de segmentación de MRI
Dipy: es una librería externa pero compatible con Python, por ende, es posible instalarla y utilizarla como una herramienta para el análisis de imágenes de resonancia magnética. Esta librería realiza la segmentación de 3 tejidos principalmente mediante la formulación bayesiana. El modelo de observación (término de probabilidad) se define como una distribución gaussiana y se usa un campo aleatorio de Markov (MRF) para modelar la probabilidad a priori de patrones dependientes del contexto de diferentes tipos de tejido del cerebro. La maximización de expectativas y los modos condicionales iterados se utilizan para encontrar la solución óptima (Zhang et al., 2001).
FSL: es una librería de herramientas para análisis de imágenes fMRI, MRI y DTI del cerebro (Jenkinson et al., 2012; Woolrich et al., 2009). Se puede acceder a sus funciones tanto por interfaz gráfica (GUI) como por línea de comandos. La librería contiene un algoritmo para extracción del cerebro a partir de imágenes de resonancia, denominado BET (Herramienta de Extracción del Cerebro, por sus siglas en inglés), el método “usa un modelo deformable que evoluciona para ajustarse a la superficie cerebral por medio de la aplicación de un conjunto de modelos locales adaptativos” (Smith, 2002). El algoritmo FAST (Herramienta de segmentación automatizada del grupo de análisis FMRIB de la Universidad de Oxford, por sus siglas en inglés) de esta librería se utiliza para segmentar los diferentes tipos de tejido en imágenes 3D del cerebro utilizando un método basado en campos aleatorios de Markov y un algoritmo de maximización de esperanza asociado (Zhang et al., 2000).
2.5. Medidas de desempeño
1. Coeficiente de Dice: también conocido como índice de similitud de Sørensen se puede utilizar en el ámbito de la recuperación de información y en otras áreas. El coeficiente de dice mide la similitud entre conjuntos (Dice, 1945), de la forma indicada la ecuación (1).
donde A es un conjunto que representa la verdad fundamental y 𝐵 representa la segmentación calculada. Ambas imágenes (conjuntos) son binario con valores 0 o 1 en cada uno de sus vóxeles o píxeles en el caso 2D (Shamir et al., 2019).
2. Índice de Jaccard: el índice de Jaccard mide la similitud en términos de la relación entre la intersección y la unión de dos conjuntos de muestras (Jaccard, 1912), de la forma indicada en la ecuación (2).
3. Area Bajo la Curva (AUC) de la curva ROC: La métrica AUC es una medida útil y ampliamente utilizada para evaluar el rendimiento de modelos de clasificación binaria y de clases múltiples. Sin embargo, no tiene en cuenta la salida numérica exacta de los modelos, sino que analiza cómo la salida clasifica los casos (Van Calster et al., 2008) permitiendo representar en un único valor el rendimiento del clasificador.
donde True Positive Rate (TPR) representa las predicciones positivas correctas entre el número total de positivos y False Positive Rate (FPR) representa las predicciones positivas incorrectas entre el número total de negativos.
4. Similitud Estructural (SSIM): El SSIM es una medida de fidelidad de imagen que ha demostrado ser muy eficaz para medir la fidelidad de las señales. El enfoque SSIM fue motivado originalmente por la observación de que las imágenes naturales tienen señales altamente estructuradas con fuertes dependencias de vecindad. Estas dependencias llevan información útil sobre las estructuras de los objetos en la escena visual. El SSIM mide las distorsiones como una combinación de tres factores: pérdida de correlación, distorsión de la luminancia y distorsión del contraste (Ndajah et al., 2010).
3. Marco Experimental
3.1. Bases de datos
Se realizó una búsqueda de múltiples bases de datos de cerebro en diferentes repositorios, páginas web y desafíos relacionados con la segmentación de MRI. Principalmente, se examinaban bases de datos públicas que tuviesen gran cantidad de imágenes y heterogeneidad etaria, ya que esto garantizaría mejores resultados en el entrenamiento de la red. Finalmente, se escogieron tres bases de datos de segmentación de MRI, que se presentan a continuación:
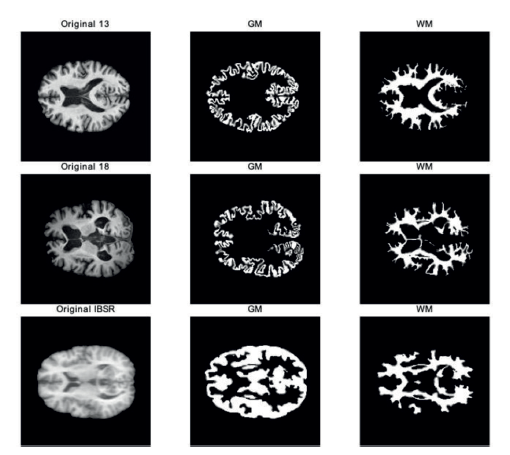
1. La primera base de datos se extrajo del desafío (A. Mendrik, 2013; A. M. Mendrik et al., 2015), la cual contiene veinte escáneres cerebrales de resonancia magnética 3T de pacientes con edades mayores a los 50 años, con diabetes y controles pareados de diversos grados de atrofia y lesiones de la sustancia blanca. Esta base de datos se divide en cinco conjuntos de datos (escaneos MRI con segmentaciones manuales) para entrenamiento y quince conjuntos de datos (solo escaneos MRI) para pruebas. En nuestro caso son de interés solamente los cinco conjuntos de datos para entrenamiento, específicamente las imágenes ponderadas en T1 de cada conjunto de datos, ya que estos contienen su propia segmentación manual realizada por un experto. Las segmentaciones manuales están constituidas por ocho etiquetas desde la 1 hasta la 8, que corresponden a un tejido diferente del cerebro, pero solo fueron de interés las etiquetas 1, 3 y 5, correspondientes a la materia gris (GM), materia blanca (WM) y fluido cerebroespinal (CSF), respectivamente. Las etiquetas adicionales se tomaron como fondo en cada una de las pruebas.
Para cada paciente, se proporcionan las siguientes secuencias:
T1_1mm: escaneo 3D ponderado en T1 (tamaño de vóxel: 1.0mm × 1.0mm × 1.0mm)
T1: escaneo 3D ponderado en T1 registrado en el T2 FLAIR (tamaño de vóxel: 0.958 mm × 0.958 mm × 3.0 mm)
T1_IR: exploración de recuperación de inversión ponderada en T1 multicorte registrada en el T2 FLAIR (tamaño de vóxel: 0.958 mm × 0.958 mm × 3.0 mm)
T2_FLAIR: escaneo FLAIR multicorte (tamaño de vóxel: 0.958 mm × 0.958 mm × 3.0 mm)
2. MRBrainS18: La segunda base de datos se extrajo del desafío (Kuijf & Bennink, 2019), la cual contiene escáneres de 30 pacientes con edades mayores a los 50 años con diabetes, demencia, Alzheimer y controles pareados (con mayor riesgo cardiovascular) de diversos grados de atrofia y lesiones de la sustancia blanca. Estos datos consisten en 7 conjuntos de imágenes de MRI cerebral. Para nuestro estudio, son de interés las imágenes ponderadas en T1 de cada conjunto de datos, ya que estos contienen su propia segmentación manual realizada por un experto. Las segmentaciones manuales están constituidas por once etiquetas desde la 0 hasta la 10, que corresponden a un tejido diferente del cerebro, pero solo fueron de interés las etiquetas 1, 3 y 5, correspondientes a GM, WM y CSF, respectivamente. Las etiquetas adicionales se tomaron como fondo en cada una de las pruebas.
Para cada paciente, se proporcionan las siguientes secuencias:
T1: Secuencia 3D ponderada en T1 (tamaño de vóxel: 0.958 mm × 0.958 mm × 3.0 mm)
T1-IR: Secuencia de recuperación de inversión ponderada en T1 de múltiples cortes (tamaño de vóxel: 0.958 mm × 0.958 mm × 3.0 mm)
T2-FLAIR: Secuencia FLAIR T2 multicorte (tamaño de vóxel: 0.958 mm × 0.958 mm × 3.0 mm)
3. Repositorio de Segmentación Cerebral de Internet (IBSR): Esta es una base de datos de MRI del cerebro que incluye segmentaciones guiadas manualmente por un experto con el fin de desarrollar y evaluar métodos de segmentación. Los volúmenes fueron puestos en el dominio público por Rohlfing (Rohlfing, 2012) y pueden obtenerse en http://www.nitrc.org/.
La base de datos incluye estudios de 18 pacientes de los cuales 4 son mujeres y 14 hombres con edades que oscilan entre 7 y 71 años. Por cada paciente se encuentran MRI de la secuencia T1 con y sin cráneo, máscara del cerebro y segmentaciones de las estructuras cerebrales. Los volúmenes tienen una resolución de 256 × 256 × 128 px y un tamaño de vóxel de 0.938 × 0.938 × 1.5 mm.
En la Figura 2 se muestra un ejemplo de cada uno de los volúmenes T1 de las bases de datos seleccionadas, en la cual se muestran también las segmentaciones manuales de los tejidos cerebrales de nuestro interés: Materia Gris (GM) y Materia Blanca (WM).
Los tres conjuntos de datos fueron estandarizados a un espacio común de dimensión 240 × 240 × 48, buscando evitar el filtrado de información que afecte la validez de los resultados obtenidos por los métodos propuestos.
3.2. Métodos
Utilizando las bases de datos descritas anteriormente, se implementó el método de segmentación propuesto, para el cual se presentaron dos variantes, una basada en representaciones bidimensionales de las imágenes (U-Net 2D) y la otra que utiliza imágenes en 3D. Estos métodos fueron comparados con dos metodologías establecidas en el estado del arte con el fin de evidenciar el progreso en la tarea de segmentación.
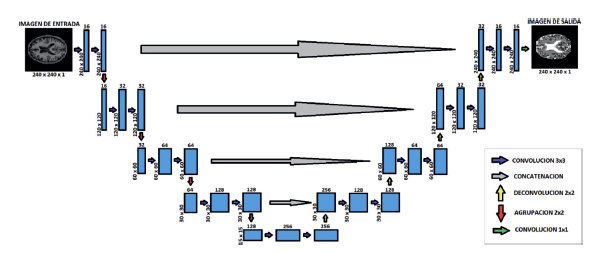
1. Segmentación empleando redes convolucionales: El primer método consiste en la creación de una red convolucional basado en la arquitectura U-Net, para segmentar imágenes de resonancia magnética en 2 y 3 dimensiones; en ambos casos se usaron para entrenamiento cuatro de las cinco MRI con segmentaciones manuales que provee el conjunto de datos MRBrainS13, la MRI restante se usará para realizar evaluación de las redes entrenadas y las técnicas de comparación. Para la U-Net 2D, en la etapa de contracción se tienen como entrada, imágenes con dimensiones de 240 × 240 × 1, haciendo referencia a que son imágenes 2D en escala de grises, seguidas de dos convoluciones de 16 filtros cada una para empezar a extraer características de la imagen. Finalmente, se aplica un MaxPooling para reducir las características similares y a su vez, reducir a la mitad el tamaño de la imagen resultante.
Luego, se aplica este mismo procedimiento de dos convoluciones y un MaxPooling para tres niveles distintos de la red, pero la variación está en que, a cada nivel siguiente, se le aplica el doble de filtros del nivel anterior en cada convolución, es decir, en el primer nivel se aplicaron 16 filtros a la imagen, luego, en el siguiente nivel se aplicarán ya no 16 sino 32 filtros, y así sucesivamente para los demás niveles hasta llegar al fondo de la red, donde se realizan 2 convoluciones de 256 filtros sin el MaxPooling para extraer características más precisas.
Después de esta última etapa de contracción, se inicia la etapa de expansión, la cual tiene como objetivo regresar la imagen a su tamaño original. Acá se aplica el proceso inverso a la convolución, es decir la convolución transpuesta, que básicamente realiza un relleno en la imagen original seguido de unas capas convoluciones, aplicando los mismos filtros de la etapa de contracción, pero de forma descendente. A su vez, después de aplicar la convolución transpuesta, a la imagen también se le concatena con la imagen correspondiente de la ruta de contracción, con el objetivo de utilizar las características extraídas allí. Este proceso mencionado anteriormente, se ejecuta para 3 niveles más de la etapa de expansión hasta el último nivel de la red, donde se realiza una última capa de convolución con un filtro de tamaño 1 × 1 para satisfacer los requisitos de predicción. El esquema completo de la red se puede apreciar en la Figura 3.
Luego de construir la red U-Net 2D, se realiza un pre-procesamiento de las imágenes de la base de datos, con la finalidad de tener imágenes estándar, con el tipo de dato correspondiente y las dimensiones correctas para proceder a realizar su entrenamiento. Para la red U-Net 3D se usaron subvolúmenes de tamaño 80 × 80 × 16, con el fin de aumentar sintéticamente el número total de datos de entrenamiento, reducir el costo computacional de la red y el número total de parámetros. Se usó la misma cantidad de filtros en cada capa que en la red U-Net 2D en su versión de tres dimensiones.
En el proceso de entrenamiento de las redes se utilizó el optimizador Adam, con tasa de aprendizaje establecida en 0.001, 𝛽 1 =0.9 y β 2 =0.999 (Kingma & Ba, 2014). La función de pérdida usada fue entropía cruzada binaria y se realizaron en total 100 épocas, almacenando la mejor época de entrenamiento basándose en el resultado de exactitud.
2. Segmentación empleando FSL: Este método se presenta como primera técnica de comparación. FSL es una librería para análisis estadístico de FMRI, MRI y DTI, en este caso, se utilizó el módulo para segmentación de tejido cerebral en materia gris (GM), materia blanca (WM) y CSF. Para la parametrización se utilizó salida por separado de las imágenes binarizadas para cada tipo de tejido, se realizó la estimación parcial del volumen, se seleccionaron 3 clases de salida correspondientes a los tejidos de interés y se utilizaron salitas tipo NIFTI. Adicionalmente, para el tipo de imagen de entrada se utilizó T1, lo que indica que se maximiza el contraste T1.
3. Segmentación empleando Dipy: Para este método se debe tener en cuenta que las imágenes de resonancia magnética deben estar sin el cráneo, de lo contrario, se deberá realizar un procesamiento adicional donde se remueva la parte no cerebral de las imágenes, esto con la finalidad de obtener mejores resultados en la segmentación.
Luego de corroborar lo anterior, se ajustan algunos parámetros importantes como el número de clases, que para este caso son 3: GM, WM y CSF. También, el factor de suavizado de la imagen denominado beta, que ayuda a eliminar puntos de ruido de estas. Este parámetro oscila entre 0 y 0.5, que para nuestro caso se definió en 0.1, ya que es donde consigue un mejor rendimiento. Otro parámetro que también se puede redefinir es el número de iteraciones, ya que por defecto este viene en 100, sin embargo, es posible que se llegue a la convergencia en menos iteraciones dependiendo del modelo, por lo que no es necesario llegar o superar dicho número de iteraciones en algunos casos, así que se determinó ajustarlo para un número total de 20 iteraciones.
Después de asignar los parámetros definidos anteriormente, se procede a crear una instancia de la clase “TissueClassifierHMRF” con su respectivo método “classify” a las imágenes de resonancia magnética, donde al final, después del número de iteraciones asignadas, se obtendrá la imagen con las etiquetas esperadas de cada tejido del cerebro.
Buscando garantizar la reproducibilidad de este estudio, los códigos para el entrenamiento y uso de los métodos propuestos y métodos base se pueden encontrar en el siguiente repositorio: https://github.com/JovianPlanet/brain_segmentation.
4. Resultados
Luego del entrenamiento de las arquitecturas de U-Net presentadas, se procedió a la evaluación tanto de los modelos propuesto como los métodos comparativos. En esta etapa, se utilizaron métricas de evaluación basadas en teoría de conjuntos, el área bajo la curva (AUC) y la SSIM que se basa en la similitud estructural de imágenes. De este modo, se muestran a continuación los resultados estadísticos y ejemplos del desempeño de los modelos para realizar segmentación tanto de materia gris como de materia blanca.
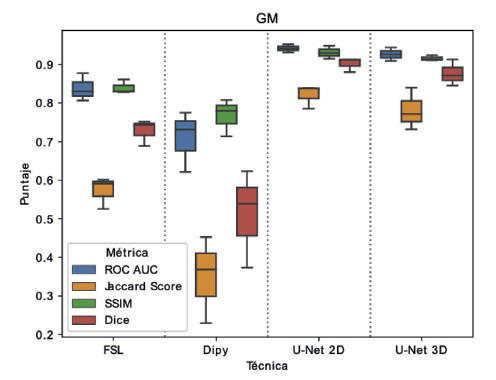
Inicialmente, en la Figura 4 se presentan los resultados obtenidos para la segmentación de materia gris usando un diagrama de caja (boxplot). Para estos resultados se muestran los desempeños evaluados con las cuatro métricas implementadas. Los resultados muestran que, para cada método de evaluación, las arquitecturas propuestas (U-Net 2D y U-Net 3D) logran alcanzar puntajes por encima de 0.9 en la mayoría de las métricas, exceptuando el puntaje de Jaccard, el cual para todos los métodos analizados presenta una caída significativa. Sin embargo, los métodos basados en U-Net para el puntaje Jaccard mantienen una mediana cercana a 0.8, mientras que para los métodos de comparación se tiene mediana inferior a 0.6 en el caso de FSL e inferior a 0.4 para Dipy. En general se puede decir que, para la segmentación de materia gris, los métodos propuestos en este trabajo mantienen una varianza inferior a los métodos con los que se compara. Además, se tiene una mediana superior y menor variación de resultados entre las diferentes métricas.
Adicionalmente, en la Figura 5 se muestran algunos resultados como ejemplo de la segmentación realizada por las técnicas propuestas y los métodos de comparación sobre un volumen del conjunto de datos MRBrainS18. En blanco se presenta el ground truth, y sobre él, utilizando transparencia, la delineación obtenida por cada método. La figura muestra que U-Net en ambas versiones consigue seguir los surcos de la estructura analizada, mientras que para las demás metodologías se marcan también ventrículos. Por otro lado, se percibe visualmente que las regiones de convergencia entre la etiqueta real y las salidas de los métodos usando U-Net son mayores que las obtenidas por FSL y Dipy. Esto se puede ver en los espacios blancos que quedan dentro de la estructura, siendo más notoria en Dipy que FSL.
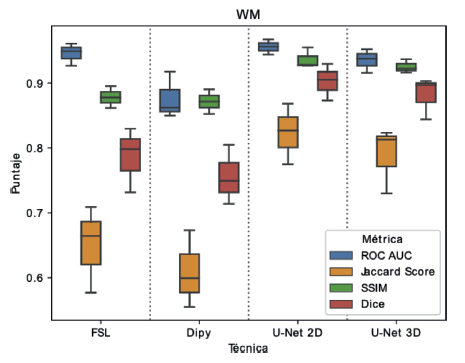
En la Figura 6 se presentan los resultados del diagrama de caja obtenidos para la segmentación de materia blanca. Estos resultados muestran a diferencia de la materia gris, una menor variabilidad entre los diferentes métodos y métricas de evaluación, encontrando que la media de los puntajes se mantiene por encima de 0.6, mientras que para materia gris podían estar por debajo de 0.4, lo cual sugiere que la segmentación de materia blanca es una tarea menos exigente que la segmentación de materia gris. Al igual que en el experimento anterior, los métodos propuestos continúan teniendo una mediana alta en el puntaje para cada métrica. En el caso del puntaje Jaccard, al igual que con la materia gris, se nota una disminución significativa de la mediana y mayor varianza en todos los métodos, aunque la caída sigue siendo menor para los métodos basados en U-Net.
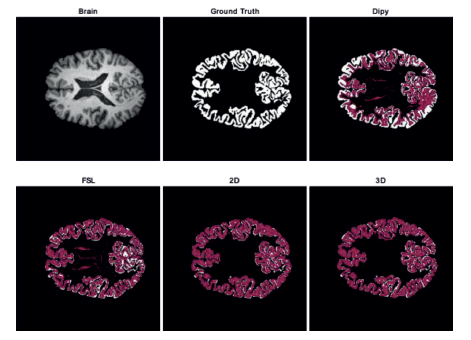
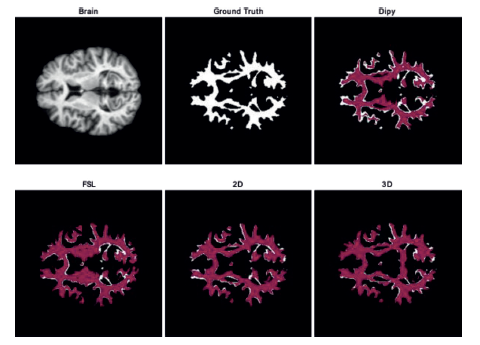
Finalmente, un ejemplo de la segmentación de la materia blanca sobre un volumen del conjunto de datos IBSR, se muestra en la Figura 7. Se presentan los resultados para las técnicas contempladas, en blanco se presenta la etiqueta de comparación (Ground truth), y sobre esta, utilizando transparencia, la delineación obtenida por cada método. En este caso los resultados son consistentes con lo reportado en el experimento anterior, en donde Dipy presenta un gran número de espacios del tejido sin segmentar y FSL a pesar de que realiza una mejor segmentación, hace marcación errónea sobre componentes que están por fuera de la estructura de interés. Por otro lado, al analizar U-Net, se puede ver como los espacios no segmentados son reducidos (similares para ambas propuestas) y además se tiene una menor marcación de áreas fuera de la estructura, en donde se puede evidenciar un mejor rendimiento cuando es utilizado el modelo 2D.
5. Conclusiones
En este trabajo se realizó la propuesta de una técnica de segmentación de tejido cerebral utilizando arquitecturas de redes neuronales, específicamente se trabajó con redes U-Net en dos versiones diferentes que recibían imágenes en 2D o volúmenes en 3D. Los resultados de esta investigación muestran como la técnica propuesta consigue mejorar el rendimiento de la tarea de segmentación de materia gris y blanca cuando se compara con metodologías recientes del estado del arte. De forma general se pudo evidenciar que las arquitecturas U-Net logran reducir significativamente la segmentación de componentes fuera de los tejidos de interés, es decir, hay una menor segmentación de los tejidos circundantes.
Al hacer la comparación entre el modelo 2D y 3D, se puede concluir que el modelo 2D es ligeramente superior al 3D cuando se observan los diagramas de cajas en todas las medidas de evaluación. Adicionalmente, al ver las áreas de segmentación, se puede ver como el modelo 2D disminuye las áreas sin segmentar para materia gris principalmente, y para materia blanca se disminuyen las segmentaciones fuera del tejido. De forma general, el modelo 2D reduce tanto los falsos positivos como falsos negativos de la segmentación.
El principal reto para que la metodología propuesta pueda ser usada en línea se encuentra en la etapa de entrenamiento, debido a la sintonización iterativa de parámetros. Posteriormente en la etapa de evaluación, la velocidad de cómputo disminuye considerablemente para ser usada en tiempo real. Sin embargo, es importante considerar que los MRI a usarse se deben llevar al espacio de entrada de las redes, es decir, 240 × 240 × 48, que permitan el uso individual en el eje axial para el modelo 2D y el uso de subvolúmenes de dimensión 80 × 80 × 16 en el modelo 3D.
Como trabajo futuro se encuentra validar el rendimiento de la arquitectura propuesta en otras estructuras cerebrales para evaluar la generalización del modelo. Por otro lado, aunque se tiene una mejora notoria en la tarea de segmentación, aún existen contornos que son difíciles de distinguir para el modelo, lo cual establece una ruta de trabaja en pro de implementar mejoras que permitan la segmentación de zonas al parecer son desafiantes para las distintas técnicas implementadas.