Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Motricidade
Print version ISSN 1646-107X
Motri. vol.11 no.4 Ribeira de Pena Dec. 2015
https://doi.org/10.6063/motricidade.3514
ARTIGO ORIGINAL
Estudo de associação entre polimorfismos no gene FTO e composição corporal em idosas brasileiras
Association study between FTO gene variations and body composition in older Brazilian women
Cláudia Cruz Lunardi1,*; Rinaldo Wellerson Pereira2; Tailce Kaley Moura Leite2; Ana Barbára Moura Siqueira2; Ricardo Moreno Lima3; Ricardo Jacó de Oliveira3
1 Universidade de Brasília, Brasília, Brasil
2 Universidade Católica de Brasília, Brasília, Brasil
3 Universidade de Brasília, Brasília, Brasil
RESUMO
Estudos de associação genética (GWAS) sugerem uma importante contribuição do gene candidato FTO na composição corporal em diferentes populações e variada faixa etária. Este estudo teve como objetivo examinar a associação entre os polimorfismos no gene FTO (rs9939609 e rs1861868) e fenótipos de adiposidade em uma amostra de idosas brasileiras não caucasianas. A amostra foi composta por 241 mulheres (66.65± 5.5 anos). Os dados referentes à composição corporal foram mensurados através da absorciometria por raios-x de dupla energia (DXA) e a genotipagem dos SNPs foi realizada utilizando o sistema SnaPShot Multiplex System (Applied Biosystems). Não foram encontradas diferenças estatisticamente significativas para as variáveis de composição corporal entre os polimorfismos estudados, mesmo após o ajuste para ancestralidade Africana. Especificamente, índice de massa corporal (p= 0.94 e p= 0.66), percentual de gordura (p= 0.42 e p= 0.93) e massa livre de gordura total (p= 0.75 e p= 0.30), sendo os valores de p referentes aos rs9939609 e rs1861868, respectivamente. Nesta amostra de idosas brasileiras os polimorfismos do gene FTO (rs9939609 e rs1861868) não apresentaram associação com fenótipos de obesidade.
Palavras-chave: genética, obesidade, idosos, mulher, densitometria, IMC, gene FTO
ABSTRACT
GenomeWide Association Studies (GWAS) suggest an important contribution of gene FTO in body composition and muscle strength in different populations and wide age. This study intended to examine the association of polymorphisms in the FTO gene (rs9939609 and rs1861868) with obesity phenotypes in a sample of Brazilian elderly women not Caucasian. The sample included 241 older women. Data concerning body composition were measured by dual energy X-ray absorptiometry (DXA) and genotyping of SNPs was carried out using the system snapshot Multiplex System (Applied Biosystems). There was no statistically significant difference for body composition variables, even after adjustment for African ancestry. Specifically, body mass index (p= 0.94 and p= 0.66), fat percentage (p= 0.42 and p= 0.93) and total fat free mass (p= 0.75 and p= 0.30) did not present significant differences between genotypes (p values related to rs9939609 and rs1861868, respectively). In this sample of Brazilian elderly, the studied FTO gene polymorphisms (rs9939609 and rs1861868) are not associated with obesity-related phenotypes.
Keywords: gene, obesity, elderly, women, densitometry, BMI, gene FTO
INTRODUÇÃO
Estudos epidemiológicos sobre o excesso de gordura corporal em idosos indicam que os distúrbios nutricionais estão associados ao aumentado risco de morbidade e de mortalidade (World Health Organization [WHO], 2000) por se relacionar a condições desfavoráveis como dificuldades respiratórias, distúrbios do aparelho locomotor, alguns tipos de câncer, doenças cardiovasculares, dislipidemia, hipertensão arterial, resistência à insulina e diabetes (Kannel, Wilson, Nam, & DAgostino, 2002; Tonstad & Hjermann, 2003; WHO, 2000). A obesidade torna-se particularmente preocupante no envelhecimento, devido à perda progressiva da massa magra concomitante ao aumento da proporção de gordura corpórea (WHO, 2000). Desta forma, atenção deve ser demandada no sentido de prevenção e identificação precoce de excesso de peso uma vez que o número de idosos vem aumentando progressivamente.
A obesidade apresenta etiologia multifatorial, envolvendo fatores nutricionais, inatividade física, aspectos psicológicos (Agurs-Collins & Bouchard, 2008) e influência hereditária. Fatores genéticos estimam cerca de 40 a 70% da variação de adiposidade (Allison, Faith, & Nathan, 1996; Bouchard & Tremblay, 1997). Evidências indicam que a obesidade é causada por uma complexa interação entre genes e ambiente (Frayling et al., 2007; Loos & Bouchard, 2008) e o estudo dessa interação é importante para determinar quais indivíduos são mais propensos a desenvolver obesidade em resposta a um determinado meio (Frayling et al., 2007; Maes, Neale, & Eaves, 1997; Marti, Moreno-Aliaga, Hebebrand, & Martínez, 2004). Embora seja bem documentada que a hereditariedade contribui para determinar a adiposidade, a identificação de genes específicos requer futuros estudos. Mais de 253 genes já foram associados com índices de adiposidade em estudos prévios, porém, apenas 61 foram reproduzidos em pelo menos cinco estudos (Rankinen et al., 2006).
Dentre os genes identificados e associados a obesidade, destaca-se o Fat Mass and Obesity Gene (FTO). O gene vem sendo associado a traços de obesidade em populações de diferentes etnias e idades (Dina et al., 2007; Frayling et al., 2007; Loos & Bouchard, 2008; Scuteri et al., 2007). Dentre os polimorfismos de nucleotídeo simples (SNP) do gene FTO, o rs9939609 é estudado com maior frequência, pois apresenta maior sucesso na taxa de genotipagem (100%) e possibilita associações com menor chances de erro (Frayling et al., 2007). Estudos mais recentes apontam uma relação direta entre o rs1861868, índice de massa corporal (IMC) e obesidade (Rampersaud et al., 2008; Rodríguez-López et al., 2010).
Os mecanismos de ação do FTO na massa corporal ainda não são claros. Há estudos que mostram que o gene age no controle da homeostase energética, atuando como regulador primário do acúmulo de gordura corporal e na regulação do jejum e leptina (Dina et al., 2007; Frayling et al., 2007; Fredriksson et al., 2008; Gerken et al., 2007). O gene foi encontrado no tecido adiposo, pâncreas, fígado (Stratigopoulos et al., 2008), musculatura esquelética estriada e cardíaca, rins, gônadas (Gerken et al., 2007; Stratigopoulos et al., 2008). Outros estudos indicam que o FTO tem efeito especialmente na adiposidade geral corporal (Freathy et al., 2008) e em particular na massa gorda subcutânea (Peeters et al., 2008).
Não obstante, a literatura disponível é controversa em relação ao gene FTO, pois embora alguns estudos tenham observado associação (Frayling et al., 2007; Liu et al., 2010), outros não confirmaram esses resultados (Jacobsson et al., 2009, 2011; Ramos, Casanova, Maturana, & Spritzer, 2011). Nesse sentido, poucos estudos foram conduzidos em indivíduos idosos e limitaram-se a utilização do IMC como método de avaliação da composição corporal. Desta forma, torna-se importante que futuros estudos avaliem a composição corporal por meio de métodos diretos como a absortometria por raios-x de dupla energia (DXA), considerando fatores que podem interferir na adiposidade como os anos de menopausa, reposição hormonal, suplementação de cálcio e níveis de atividade física. Ademais, tendo conhecimento da alta miscigenação da população brasileira e das possíveis complicações da cor de pele auto referida em estudos de associação (Lins et al., 2011), 19 polimorfismos informativos de ancestralidade foram genotipados e considerados como covariáveis nas análises. Uma vez que o número de idosos vem aumentando consistentemente e que o percentual de gordura aumenta com o avançar da idade, é importante investigar genes candidatos nessa população.
Dessa forma, o presente estudo buscou examinar os genes candidatos previamente identificados em estudos de GWAS para fenótipos de obesidade. Especificamente, o objetivo foi replicar os resultados de estudos anteriores, para verificar se existe uma associação entre polimorfismos no gene FTO (rs9939609 e rs1861868) e obesidade em uma amostra de mulheres idosas brasileiras.
MÉTODOS
Amostra
As voluntárias do estudo tinham idade entre 60 anos e 85 anos e foram convidadas a ingressar no presente estudo através de ligações telefônicas, nas quais era exposto um breve panorama da investigação. Inicialmente cerca de 500 ligações foram realizadas das quais 300 aceitaram em participar. As principais causas que levaram à falta de êxito dos demais convites incluíram alteração do número telefônico, doença e falta de interesse. Após aplicação dos critérios de exclusão, um total de 241 voluntárias (idade média de 66.65 ± 5.5 anos) compuseram a amostra.
Foram excluídas do estudo as participantes que: a) não tinham nacionalidade brasileira, b) não foram capazes de caminhar de forma independente, c) possuíam prótese de quadril unilateral ou bilateral d) possuíam prótese metálica, e) fumantes, f) apresentavam desordem metabólica ou endócrina que sabidamente afeta o sistema muscular; g) apresentavam anormalidade de condução ou perfusão cardíaca que contraindique a prática de atividades físicas. Para participar as voluntárias do estudo assinaram um termo de consentimento escrito. O estudo foi aprovado pelo Comitê de Ética da Universidade (CEP/UCB 014/2007).
As idosas informaram os anos de menopausa, uso de reposição hormonal (se sim ou não) e uso de suplemento de cálcio (se sim ou não).
Instrumentos e Procedimentos
Avaliação da Composição Corporal
Para realização das medidas antropométricas as participantes estavam descalças e vestindo roupas leves. A massa corporal foi mensurada em uma balança digital calibrada digital (mod 2006pp TOLEDO, Brasil, resolução de 0.10 kg). Para mensuração da estatura utilizou-se um estadiômetro (CARDIOMED, Brasil, precisão de 0.10 cm) fixado na parede.
Os dados referentes à Massa Livre de Gordura (MLG) foram mensurados através da absortometria por raios-x de dupla energia (DXA). Para tal, utilizou-se o equipamento da marca Lunnar, modelo DPX-IQ (Lunar Corporation, Madison, WI, USA) conforme procedimentos previamente especificados por Moreno Lima et al. (2007). Brevemente, as voluntárias foram posicionadas em decúbito dorsal sobre a mesa do equipamento, sendo em seguida cuidadosamente posicionadas de forma que ficassem totalmente centralizadas em relação às laterais da mesa. Após análise de toda área corporal, o DXA possibilita a determinação da MLG para diferentes regiões corporais. Com base nesses resultados, é possível se chegar ao valor da MLG Apendicular (MLGA), a qual é representada pelo somatório da MLG dos membros inferiores e superiores.
Avaliação do nível de atividade física
Para verificar os níveis habituais de atividade física de cada uma das participantes, foi utilizada a versão longa do IPAQ (do inglês, International Physical Activity Questionaire). O questionário foi administrado em entrevistas face a face, conforme recomendação de uso em países em desenvolvimento. O IPAQ foi desenvolvido como um instrumento para monitorar, de forma padronizada, a atividade e inatividade física em diversos países do Mundo (Craig et al., 2003), tendo a Organização Mundial de Saúde provido o devido suporte. O modelo usado no presente estudo foi a tradução oficial em português (disponível no site www.celafiscs.com.br), previamente validada para a população brasileira. A avaliação leva em consideração a duração e frequência das atividades físicas realizadas em uma semana, considerando-se apenas sessões superiores a 10 minutos contínuos. Os resultados do questionário possibilitam a divisão em quatro categorias: sedentários, insuficientemente ativos, ativos e muito ativos.
Genotipagem
Amostra sanguínea de todas as participantes foi coletada na veia antecubital e o DNA genômico de alto peso molecular foi extraído dos leucócitos periféricos pelo método Salting Out (Miller). A genotipagem dos SNPs foi realizada utilizando o sistema SnaPShot Multiplex System (Applied Biosystems). A genotipagem através deste sistema envolveu a amplificação pela PCR dos fragmentos de DNA contendo os sítios polimórficos. As condições da PCR obedeceram a seguinte variação de temperatura: 05 minutos a 95º; 14 ciclos (01 minuto 95º - 01 minuto a 63º - 01 minuto a 72º); 25 ciclos (01 minuto a 95º - 01 minuto a 56º - 01 minuto a 72º); 10 minutos a 72º; e mantido em 10º até a reação ser retirada do termociclador. As sequencias de primers foreward e reverse, respectivamente, utilizadas para o SNP rs9939609 foram 5–CTTGCGACTGCTGTGAATTT–3 e 5– GCCCAAGGATGGTGTTTCTA–3. Para o SNP rs1861868 utilizaram-se as sequencias de primers 5–CGCATCTCTGCAACTCTTTT–3 para foreward e 5– CCCCTTGCATCAGAGTGTTT– 3 para reverse. Em seguida a purificação dos produtos da amplificação com ExoI e SAP, respectivamente uma exonuclease e uma fosfatase para eliminação dos primers e do excesso de dNTPs utilizados na PCR. Os produtos da PCR de diferentes fragmentos contendo até dez SNPs a serem genotipados foram misturados. Uma alíquota desta mistura foi adicionada ao kit e a outra mistura contendo os primers adjacentes aos SNPs a serem genotipados. Esta mistura foi levada ao termociclador onde utilizando um programa de ciclagem recomendado pelo fabricante permite-se a extensão de uma única base. A cada um dos ddNTPs tem-se uma fluorescência diferente o que permitiu a genotipagem dos SNPs. Os fragmentos gerados durante a reação para extensão de uma única base foram purificados com SAP para remoção do excesso de ddNTPs fluorescentes não incorporados. Uma alíquota deste produto purificado foi adicionada a um padrão de tamanho molecular (GS120 Liz, Applied Biosystems) e a formamida altamente deionizada. Esta mistura foi desnaturada a 95oC durante 3 minutos e em seguida mantida em gelo por no mínimo 5 minutos. As amostras desnaturadas foram submetidas à eletroforese em um seqüenciador automático de DNA ABI Prism 3100. Os eletroferogramas foram analisados pelo software GeneMapper v3.5. A genotipagem de 19 marcadores informativos de ancestralidade foi realizada sob as mesmas condições descritas acima.
Para estimar a ancestralidade genômica 19 marcadores informativos de ancestralidade foram genotipados para todas as amostras. A ancestralidade em cada população foi estimada utilizando o software ADMIXMAP (http://homepages.ed.ac.uk/pmckeigu/admixmap/), com 2.500 iterações para o período de burnin e 10.000 iterações para medir dados de parâmetro.
ANÁLISE ESTATÍSTICA
Para verificar a normalidade da distribuição dos dados foi utilizado o teste de Kolmogorov-Smirnov. Os dados são apresentados através da estatística descritiva, utilizando-se os procedimentos de média e erro padrão. Para verificar se a freqüência alélica das voluntárias encontrava-se de acordo com o equilíbrio de Hardy-Weinberg, foi utilizado o teste chi-quadrado. Foi utilizada a análise de variância (ANOVA) para testar a existência de diferenças entre os genótipos nas seguintes variáveis: idade, massa corporal, estatura, IMC e percentual de gordura. Para verificar a existência de diferenças entre os genótipos para as variáveis não contínuas como suplementação de cálcio, uso de reposição hormonal e nível de atividade física, foi conduzido o teste chi-quadrado. Para verificar a associação entre os genótipos com os fenótipos relacionados à composição corporal, foram realizados modelos de análise de covariância (ANCOVA). Ocorrendo diferença significativa em alguma das variáveis, testes de comparações múltiplas LSD foram adotados para identificação de contrastes relevantes entre as médias. Na ANCOVA utilizou-se como covariáveis os anos de menopausa, reposição hormonal, suplementação de cálcio, níveis de atividade física e ancestralidade africana.
O nível de significância adotado foi de p≤ 0.05 e os dados foram analisados por meio do programa SPSS 13.0 for Windows.
RESULTADOS
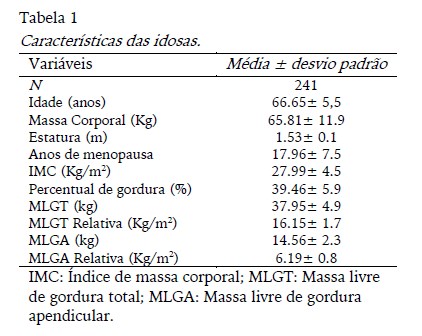
Os dados descritivos da amostra (idade, anos de menopausa e composição corporal) estão apresentados na tabela 1. De acordo com Organização Mundial da Saúde (WHO, 2000) 73.40% das idosas foram classificadas como excesso de peso (sobrepeso ou obesidade - IMC> 25kg/m²). Em relação à reposição hormonal e suplementação de cálcio, 19 (7.90%) e 56 (23.70%) mulheres estavam em uso de ambos, respetivamente. Os níveis de atividade física foram os seguintes: cinco (2.10%) eram sedentárias, 69 (28.60%) eram insuficientemente ativas, 163 (67.60%) eram ativas e quatro (1.70%) muito ativas.
As distribuições genotípicas do gene FTO (rs9939609 e rs1861868) estiveram de acordo com equilíbrio de Hardy-Weingerg (p> 0.05). Os resultados da composição corporal de acordo com o genótipo do rs9939609 estão apresentados na Tabela 2. A frequência dos alelos A e T foram 0.39 e 0.61, respectivamente. A prevalência de excesso de peso foi de 69.20%, 77.80% e 70.20%, respectivamente, para os genótipos A/A, A/T e T/T, no entanto, o teste chi-quadrado não revelou diferença estatisticamente significativas entre os grupos. Além disso, a ANOVA e ANCOVA não revelaram diferença estatística significante para idade, IMC e composição corporal avaliada pela DXA.
A Tabela 3 apresenta os resultados da composição corporal de acordo com o genótipo rs1861868. Não foram observadas diferenças significativas para rs1861868 nas variáveis estudadas. A frequência do alelo G e A foi 0.47 e 0.52, respectivamente. A prevalência de excesso de peso foi 74.50%, 75.60% e 68.70%, respectivamente, para os genótipos G/G, G/A e A/A, no entanto, o teste chi-quadrado não revelou diferenças significativas. Da mesma forma, os testes estatísticos ANOVA e ANCOVA não apontaram diferença estatística significante para idade, IMC e composição corporal.
DISCUSSÃO
Com base em resultados de GWAS prévios apontando uma associação entre polimorfismos no gene FTO (rs9939609 e rs1861868) e obesidade em populações diferentes com uma ampla faixa etária, o presente estudo objetivou verificar essa associação em uma amostra brasileira. Entretanto, esse estudo torna-se importante pois envolveu uma amostra de idosas e avaliou, além do IMC, a adiposidade por um método mais sofisticado que é o DXA. Os principais resultados não indicaram associação entre as duas variantes genéticas examinadas com nenhuma das variáveis relacionadas a gordura corporal (i.e., massa corporal, IMC, massa livre de gordura ou percentual de gordura).
O primeiro GWAS que identificou a relação entre o gene FTO e a obesidade data de 2007 (Frayling et al., 2007). No artigo em questão identificou-se múltiplos polimorfismos do gene associados a diabetes mellitus do tipo 2 (DM2). No entanto, após ajuste para o IMC a associação não foi verificada, mostrando que, possivelmente, os polimorfismos estavam associados ao IMC e se portou como um fator de risco para o desenvolvimento de DM2. Carreadores do genótipo A/A apresentavam, aproximadamente, uma média de 3kg a mais na massa corporal e uma probabilidade de 1.67 vezes de apresentar obesidade enquanto o genótipo heterozigoto apresentou um ganho de 1.50 kg (Frayling et al., 2007).
Frayling et al. (2007) foi o pioneiro, no entanto, os estudos de replicação se expandiram para outras etnias, idades e raças (Dina et al., 2007; Loos & Bouchard, 2008; Scuteri et al., 2007). Recentemente, Mei et al. (2012) concluíram que as variações do gene FTO (em especial o alelo A) podem acelerar o início precoce e aumentar a probabilidade de sobrepeso em adultos jovens. Resultados semelhantes foram apontados por Pyrzak, Wisniewska, Majcher, Tysarowski e Demkow (2013) ao estudar o polimorfismo rs9939609 do gene FTO. Os autores concluíram que esse polimorfismo esta associado a IMC e %G em crianças.
O presente estudo não observou diferença estatística significante para os polimorfismos candidatos do gene FTO, mesmo após análises adicionais com covariáveis (idade, anos de menopausa, nível de atividade física, reposição hormonal, suplementação de cálcio e ancestralidade africana) nas variáveis relacionadas ao fenótipo de massa gorda (IMC, %G e prevalência de excesso de peso). Esses resultados corroboram os estudos realizados por Jacobsson et al. (2009), Ramos, Casanova, Maturana, e Spritzer (2011) e Jacobsson et al. (2011). Jacobsson et al. (2009) verificaram que o alelo de risco (A - rs9939609) para obesidade não se associa ao aumento do IMC em homens adultos suecos.
Ramos et al. (2011) testaram a associação entre o rs9939609 e variáveis metabólicas e cardiovasculares no período pós-menopausa em mulheres brasileiras e concluíram que o polimorfismo é um preditor para doenças cardiovasculares (valores elevados de razão cintura-quadril, pressão arterial e acumulação lipídica), no entanto, não verificaram associação do SNP com o IMC. Após a revisão de literatura realizada, verificou-se que este é o único estudo a examinar o gene FTO em brasileiros idosos. Jacobsson et al. (2011), concluíram que o gene FTO é menos associado a sobrepeso e fenótipos de composição corporal (circunferência da cintura, massa gorda total, massa gorda do tronco, massa gorda dos membros inferiores, tecido visceral e subcutâneo) em idosos do que em jovens.
Schousboe et al. (2004) e Coady et al. (2002) sugerem que a influência genética na obesidade e massa de gordura diminui com o aumento da idade em função da interferência do estilo de vida. As variações no gene FTO podem influenciar a ingestão de alimentos causando o aumento do peso corporal (Speakman, Rance, & Johnstone, 2008; Wardle, Llewellyn, Sanderson, & Plomin, 2009). Os portadores de pelo menos uma cópia deste SNP escolhem com maior frequência alimentos mais palatáveis e mais calóricos e mostram sinais de redução da percepção da saciedade (den Hoed, Westerterp-Plantenga, Bouwman, Mariman, & Westerterp, 2009).
A amostra do estudo de Jacobsson et al. (2011) foi composta por uma idade média de 70.20 anos, a qual é similar a deste trabalho (66.50 anos) e Ramos et al. (2011) estudaram mulheres em período pós menopausa (idade media de 52 anos), como a amostra desse estudo. As demais pesquisas que, diferentemente, encontraram associação entre o rs9939609 e fenótipos ligados à obesidade (Frayling et al., 2007; Liu et al., 2010) possuem médias de idade diferentes e amplitude de idades elevadas. Fatos que podem causar viés de análise, além de que, aparentemente, os efeitos do FTO na composição corporal parecem menos profundos entre idosos comparados com idade mais jovens (Jacobsson et al., 2011).
Outro polimorfismo do gene FTO associado a fenótipos de massa gorda é rs1861868. Ao realizar a busca deste polimorfismo no Banco de Dados de Polimorfismo de Nucleotídeo Único verifica-se a citação do polimorfismo em apenas 3 artigos, sendo um destes de revisão. Rampersaud et al. (2008) identificou que o SNP rs1861868 está associado ao IMC de indivíduos que apresentaram baixos escores para nível de atividade física (após ajuste para sexo e idade), não revelando efeito naqueles que apresentaram nível de atividade física acima da média (gasto energético acima de 860 e 980 calorias para mulheres e homens, respectivamente). Rodríguez-López et al. (2010) identificaram que os indivíduos portadores do alelo G para rs1861868 mostraram um aumento do risco (odds ratio de 3.03; intervalo de confiança de 95%: 1.74 – 5.27) a desenvolver obesidade mórbida. No presente estudo, não foi verificado diferença estatística significante entre os genótipos (rs1861868).
Algumas limitações da presente investigação precisam ser consideradas. A amostra foi composta por Brasileiras, uma população que apresenta um alto grau de miscigenação. Dessa forma, pode ser argumentado que a não existência de associação foi influenciada pela ancestralidade genômica. Nesse sentido, a genotipagem de polimorfismos informativos de ancestralidade e a consideração da ancestralidade genética estimada como covariável não alterou a associação observada em um estudo prévio do nosso grupo (Moreno Lima et al., 2007). Outra limitação do estudo refere-se a falta de mensuração do perímetro da cintura, optando-se pela utilização apenas do DXA. Em adendo, as participantes foram exclusivamente mulheres, o que isola a bem conhecida influência do gênero em associações genéticas com fenótipos relacionados ao músculo-esquelético.
Possivelmente, diversos são os genes que contribuem para fenótipos relacionados à adiposidade. A identificação de polimorfismos funcionais que contribuem para os fenótipos de adiposidade aumentará o conhecimento dos mecanismos fisiológicos que permeiam a obesidade. A combinação da informação genética com os fatores de risco ambientais pode ser futuramente utilizada para identificar aqueles com risco aumentado e encaminhá-los a programas de prevenção e tratamento.
Por outro lado, a identificação desses genes é uma tarefa desafiadora, particularmente, devido ao fato de que outro polimorfismo pode ser uma variável de confundimento para aquele polimorfismo que está sendo estudado (Thompson et al., 2004). Resultados conflituantes não são inesperados em estudos de associação, podendo estar relacionado às diferenças de etnia, interações com outros genes ou a definição do fenótipo (Zmuda, Cauley, & Ferrell, 2000). Outros fatores de confundimento incluem diferenças entre os genótipos para idade e hábitos que interfiram no fenótipo estudado, bem como o método utilizado para avaliar a variável de estudo.
CONCLUSÕES
Com base nos resultados observados, conclui-se que o gene FTO não é determinante de fenótipos relacionados à adiposidade em uma amostra de mulheres idosas brasileiras. A deteção de variações genéticas associadas à obesidade poderá ser útil na identificação de indivíduos predispostos e implementação precoce de estratégias preventivas. Entretanto, provavelmente diversos são os genes que interagem entre si e com o meio para determinar fenótipos complexos e embora a hereditariedade pareça contribuir para a adiposidade corporal, alterações no estilo de vida constituem conduta vital no combate a obesidade. Futuros estudos são necessários para replicar os resultados apresentados e elucidar o papel do gene FTO na determinação de fenótipos relacionados à adiposidade em mulheres idosas.
REFERÊNCIAS
Agurs-Collins, T., & Bouchard, C. (2008). Gene-Nutrition and Gene-Physical Activity Interactions in the Etiology of Obesity. Obesity, 16(Suppl 3), S2–S4. http://doi.org/10.1038/oby.2008.510 [ Links ]
Allison, D. B., Faith, M. S., & Nathan, J. S. (1996). Rischs lambda values for human obesity. International Journal of Obesity and Related Metabolic Disorders, 20(11), 990–999. [ Links ]
Bouchard, C., & Tremblay, A. (1997). Genetic influences on the response of body fat and fat distribution to positive and negative energy balances in human identical twins. The Journal of Nutrition, 127(5 Suppl), 943S–947S. [ Links ]
Coady, S. A., Jaquish, C. E., Fabsitz, R. R., Larson, M. G., Cupples, L. A., & Myers, R. H. (2002). Genetic variability of adult body mass index: a longitudinal assessment in framingham families. Obesity Research, 10(7), 675–681. http://doi.org/10.1038/oby.2002.91 [ Links ]
Craig, C. L., Marshall, A. L., Sjöström, M., Bauman, A. E., Booth, M. L., Ainsworth, B. E., Oja, P. (2003). International physical activity questionnaire: 12-country reliability and validity. Medicine and Science in Sports and Exercise, 35(8), 1381–1395. http://doi.org/10.1249/01.MSS.0000078924.61453.FB [ Links ]
den Hoed, M., Westerterp-Plantenga, M. S., Bouwman, F. G., Mariman, E. C. M., & Westerterp, K. R. (2009). Postprandial responses in hunger and satiety are associated with the rs9939609 single nucleotide polymorphism in FTO. The American Journal of Clinical Nutrition, 90(5), 1426–1432. http://doi.org/10.3945/ajcn.2009.28053 [ Links ]
Dina, C., Meyre, D., Gallina, S., Durand, E., Körner, A., Jacobson, P., Froguel, P. (2007). Variation in FTO contributes to childhood obesity and severe adult obesity. Nature Genetics, 39(6), 724–726. http://doi.org/10.1038/ng2048 [ Links ]
Frayling, T. M., Timpson, N. J., Weedon, M. N., Zeggini, E., Freathy, R. M., Lindgren, C. M., McCarthy, M. I. (2007). A common variant in the FTO gene is associated with body mass index and predisposes to childhood and adult obesity. Science (New York, N.Y.), 316(5826), 889–894. http://doi.org/10.1126/science.1141634 [ Links ]
Freathy, R. M., Timpson, N. J., Lawlor, D. A., Pouta, A., Ben-Shlomo, Y., Ruokonen, A., Frayling, T. M. (2008). Common variation in the FTO gene alters diabetes-related metabolic traits to the extent expected given its effect on BMI. Diabetes, 57(5), 1419–1426. http://doi.org/10.2337/db07-1466 [ Links ]
Fredriksson, R., Hägglund, M., Olszewski, P. K., Stephansson, O., Jacobsson, J. A., Olszewska, A. M., Schiöth, H. B. (2008). The obesity gene, FTO, is of ancient origin, up-regulated during food deprivation and expressed in neurons of feeding-related nuclei of the brain. Endocrinology, 149(5), 2062–2071. http://doi.org/10.1210/en.2007-1457 [ Links ]
Gerken, T., Girard, C. A., Tung, Y.-C. L., Webby, C. J., Saudek, V., Hewitson, K. S., Schofield, C. J. (2007). The obesity-associated FTO gene encodes a 2-oxoglutarate-dependent nucleic acid demethylase. Science (New York, N.Y.), 318(5855), 1469–1472. http://doi.org/10.1126/science.1151710 [ Links ]
Jacobsson, J. A., Almén, M. S., Benedict, C., Hedberg, L. A., Michaëlsson, K., Brooks, S., Schiöth, H. B. (2011). Detailed analysis of variants in FTO in association with body composition in a cohort of 70-year-olds suggests a weakened effect among elderly. PloS One, 6(5), e20158. http://doi.org/10.1371/journal.pone.0020158 [ Links ]
Jacobsson, J. A., Risérus, U., Axelsson, T., Lannfelt, L., Schiöth, H. B., & Fredriksson, R. (2009). The common FTO variant rs9939609 is not associated with BMI in a longitudinal study on a cohort of Swedish men born 1920-1924. BMC Medical Genetics, 10, 131. http://doi.org/10.1186/1471-2350-10-131 [ Links ]
Kannel, W. B., Wilson, P. W. F., Nam, B.-H. o, & DAgostino, R. B. (2002). Risk stratification of obesity as a coronary risk factor. American Journal of Cardiology, 90(7), 697–701. http://doi.org/10.1016/S0002-9149(02)02592-4 [ Links ]
Lins, T. C., Vieira, R. G., Abreu, B. S., Gentil, P., Moreno-Lima, R., Oliveira, R. J., & Pereira, R. W. (2011). Genetic heterogeneity of self-reported ancestry groups in an admixed Brazilian population. Journal of Epidemiology / Japan Epidemiological Association, 21(4), 240–245. [ Links ]
Liu, G., Zhu, H., Lagou, V., Gutin, B., Stallmann-Jorgensen, I. S., Treiber, F. A., Snieder, H. (2010). FTO variant rs9939609 is associated with body mass index and waist circumference, but not with energy intake or physical activity in European- and African-American youth. BMC Medical Genetics, 11, 57. http://doi.org/10.1186/1471-2350-11-57 [ Links ]
Loos, R. J. F., & Bouchard, C. (2008). FTO: the first gene contributing to common forms of human obesity. Obesity Reviews, 9(3), 246–250. http://doi.org/10.1111/j.1467-789X.2008.00481.x [ Links ]
Maes, H. H., Neale, M. C., & Eaves, L. J. (1997). Genetic and environmental factors in relative body weight and human adiposity. Behavior Genetics, 27(4), 325–351. [ Links ]
Marti, A., Moreno-Aliaga, M. J., Hebebrand, J., & Martínez, J. A. (2004). Genes, lifestyles and obesity. International Journal of Obesity, 28(S3), S29–S36. http://doi.org/10.1038/sj.ijo.0802808 [ Links ]
Mei, H., Chen, W., Mills, K., He, J., Srinivasan, S. R., Schork, N., Berenson, G. S. (2012). Influences of FTO gene on onset age of adult overweight. Human Genetics, 131(12), 1851–1859. http://doi.org/10.1007/s00439-012-1204-0 [ Links ]
Moreno Lima, R., Silva de Abreu, B., Gentil, P., Cesar de Lima Lins, T., Grattapaglia, D., Pereira, R. W., & Jacó de Oliveira, R. (2007). Lack of association between vitamin D receptor genotypes and haplotypes with fat-free mass in postmenopausal Brazilian women. The Journals of Gerontology. Series A, Biological Sciences and Medical Sciences, 62(9), 966–972. [ Links ]
Peeters, A., Beckers, S., Verrijken, A., Roevens, P., Peeters, P., Van Gaal, L., & Van Hul, W. (2008). Variants in the FTO gene are associated with common obesity in the Belgian population. Molecular Genetics and Metabolism, 93(4), 481–484. http://doi.org/10.1016/j.ymgme.2007.10.011 [ Links ]
Pyrzak, B., Wisniewska, A., Majcher, A., Tysarowski, A., & Demkow, U. (2013). Relation of fat-mass and obesity-associated gene polymorphism to fat mass content and body mass index in obese children. Advances in Experimental Medicine and Biology, 756, 255–262. http://doi.org/10.1007/978-94-007-4549-0_32 [ Links ]
Ramos, R. B., Casanova, G. K., Maturana, M. A., & Spritzer, P. M. (2011). Variations in the fat mass and obesity-associated (FTO) gene are related to glucose levels and higher lipid accumulation product in postmenopausal women from southern Brazil. Fertility and Sterility, 96(4), 974–979. http://doi.org/10.1016/j.fertnstert.2011.07.1148 [ Links ]
Rampersaud, E., Mitchell, B. D., Pollin, T. I., Fu, M., Shen, H., OConnell, J. R., Snitker, S. (2008). Physical activity and the association of common FTO gene variants with body mass index and obesity. Archives of Internal Medicine, 168(16), 1791–1797. http://doi.org/10.1001/archinte.168.16.1791 [ Links ]
Rankinen, T., Zuberi, A., Chagnon, Y. C., Weisnagel, S. J., Argyropoulos, G., Walts, B., Bouchard, C. (2006). The human obesity gene map: the 2005 update. Obesity, 14(4), 529–644. http://doi.org/10.1038/oby.2006.71 [ Links ]
Rodríguez-López, R., González-Carpio, M., Serrano, M. V., Torres, G., García de Cáceres, M. T., Herrera, T., Luengo, L. M. (2010). [Association of FTO gene polymorphisms and morbid obesity in the population of Extremadura (Spain)]. Endocrinología Y Nutrición: Órgano De La Sociedad Española De Endocrinología Y Nutrición, 57(5), 203–209. http://doi.org/10.1016/j.endonu.2010.03.002 [ Links ]
Schousboe, K., Visscher, P. M., Erbas, B., Kyvik, K. O., Hopper, J. L., Henriksen, J. E., Sørensen, T. I. A. (2004). Twin study of genetic and environmental influences on adult body size, shape, and composition. International Journal of Obesity and Related Metabolic Disorders: Journal of the International Association for the Study of Obesity, 28(1), 39–48. http://doi.org/10.1038/sj.ijo.0802524 [ Links ]
Scuteri, A., Sanna, S., Chen, W.-M., Uda, M., Albai, G., Strait, J., Abecasis, G. R. (2007). Genome-wide association scan shows genetic variants in the FTO gene are associated with obesity-related traits. PLoS Genetics, 3(7), e115. http://doi.org/10.1371/journal.pgen.0030115 [ Links ]
Speakman, J. R., Rance, K. A., & Johnstone, A. M. (2008). Polymorphisms of the FTO gene are associated with variation in energy intake, but not energy expenditure. Obesity, 16(8), 1961–1965. http://doi.org/10.1038/oby.2008.318 [ Links ]
Stratigopoulos, G., Padilla, S. L., LeDuc, C. A., Watson, E., Hattersley, A. T., McCarthy, M. I., Leibel, R. L. (2008). Regulation of Fto/Ftm gene expression in mice and humans. American Journal of Physiology. Regulatory, Integrative and Comparative Physiology, 294(4), R1185-1196. http://doi.org/10.1152/ajpregu.00839.2007 [ Links ]
Thompson, P. D., Moyna, N., Seip, R., Price, T., Clarkson, P., Angelopoulos, T., Hoffman, E. P. (2004). Functional polymorphisms associated with human muscle size and strength. Medicine and Science in Sports and Exercise, 36(7), 1132–1139. [ Links ]
Tonstad, S., & Hjermann, I. (2003). A high risk score for coronary heart disease is associated with the metabolic syndrome in 40-year-old men and women. European Journal of Cardiovascular Risk, 10(2), 129–135. http://doi.org/10.1177/174182670301000208 [ Links ]
Wardle, J., Llewellyn, C., Sanderson, S., & Plomin, R. (2009). The FTO gene and measured food intake in children. International Journal of Obesity (2005), 33(1), 42–45. http://doi.org/10.1038/ijo.2008.174 [ Links ]
WHO Consultation on Obesity, & World Health Organization. (2000). Obesity : preventing and managing the global epidemic : report of a WHO consultation. Geneva: World Health Organization. Obtido de http://apps.who.int/iris/handle/10665/42330 [ Links ]
Zmuda, J. M., Cauley, J. A., & Ferrell, R. E. (2000). Molecular epidemiology of vitamin D receptor gene variants. Epidemiologic Reviews, 22(2), 203–217. [ Links ]
Agradecimentos:
Os autores agradecem às voluntárias do estudo.
Conflito de Interesses:
Nada a declarar.
Financiamento:
Fundo de Apoio a Pesquisa do Distrito Federal (FAP-DF - 193.000.555/2009) e Conselho Nacional de Desenvolvimento Cientifico e Tecnológico (487622/2012-0).
Artigo recebido a 22.02.2014; Aceite a 16.03.2015
*Autor correspondente: SMU,QRS,RCG, Rua E, Brasília, Brasil - E-mail: claudia_lunardi@yahoo.com.br